FARMASÖTİK KİMYADA SANAL TARAMA
Sanal tarama ( Virtual Screening ), bir ilaç hedefine, tipik olarak bir protein reseptörüne veya enzime bağlanma olasılığı en yüksek olan yapıları tanımlamak amacıyla, küçük moleküllerin kütüphanelerini aramak için ilaç keşfinde kullanılan bir hesaplama tekniğidir. [2] [3] Sanal tarama, bilgisayar programları kullanılarak “çok büyük bileşik kütüphanelerinin otomatik olarak değerlendirilmesi” olarak tanımlanmıştır. [4] Bu tanımdan da anlaşılacağı üzere, VS büyük ölçüde 1060 ‘ın üzerinde akla yatkın bileşiğin muazzam kimyasal alanının nasıl olduğuna odaklanan bir sayı oyunu olmuştur. [5]
Tüm kimyasal evreni araştırmak teorik olarak ilginç bir sorun olabilse de, daha pratik VS senaryoları, hedeflenen kombinatoryal kütüphanelerin tasarlanması ve optimize edilmesine ve şirket içi bileşik depolarından veya satıcı tekliflerinden mevcut bileşiklerin kütüphanelerinin zenginleştirilmesine odaklanmaktadır. Yöntemin doğruluğu arttıkça, sanal tarama ilaç keşif sürecinin ayrılmaz bir parçası haline gelmiştir . [6] [1] Sanal Tarama, tarama için kurum içi veritabanı bileşiklerini seçmek, harici olarak satın alınabilecek bileşikleri seçmek ve daha sonra hangi bileşiğin sentezlenmesi gerektiğini seçmek için kullanılabilir.
YÖNTEMLER
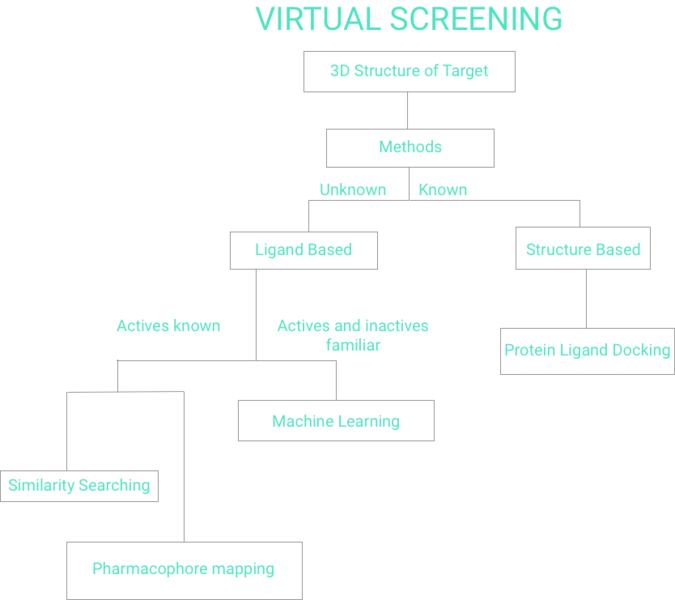
İki geniş tarama tekniği kategorisi vardır: ligand tabanlı ve yapı tabanlı. [7] Bu sayfanın geri kalanı Şekil 1 Sanal Taramanın Akış Şemasını yansıtacaktır.
LİGAND TABANLI
Bir reseptöre bağlanan bir dizi yapısal olarak çeşitli ligand göz önüne alındığında, bu tür ligand kümesinde bulunan kolektif bilgilerin kullanılmasıyla bir reseptör modeli oluşturulabilir. Bunlar farmakofor modelleri olarak bilinir. Aday bir ligand daha sonra onunla uyumlu olup olmadığını ve dolayısıyla bağlanmasının muhtemel olup olmadığını belirlemek için farmakofor modeliyle karşılaştırılabilir. [8]
Ligand tabanlı sanal taramaya başka bir yaklaşım, bir veya daha fazla aktif ligand yapısına karşı bir molekül veri tabanını taramak için 2D kimyasal benzerlik analiz yöntemleri [9] kullanmaktır.
Ligand tabanlı sanal tarama için popüler bir yaklaşım, bilinen aktiflerinkine benzer şekle sahip moleküller aramaya dayanır, çünkü bu tür moleküller hedefin bağlanma yerine uyacak ve dolayısıyla hedefi bağlayacaktır. Bu teknik sınıfının literatürde bir takım ileriye dönük uygulamaları bulunmaktadır. [10] [11] [12] Bu 3D yöntemlerin farmakoforik uzantıları da web-sunucuları olarak serbestçe temin edilebilir. [13] [14]
YAPI TABANLI
Yapıya dayalı sanal tarama, aday ligandların bir protein hedefine yerleştirilmesini ve ardından ligandın yüksek afinite ile proteine bağlanma olasılığını tahmin etmek için bir puanlama fonksiyonu uygulanmasını içerir . [15] [16] [17] Potansiyel sanal taramaya yönelik web sunucuları herkes tarafından kullanılabilir. [18] [19]
HİBRİT YÖNTEMLER
Yapısal ve ligand benzerliğine dayanan hibrit yöntemler de geleneksel VLS yaklaşımlarının sınırlamalarının üstesinden gelmek için geliştirilmiştir. Bu metodolojiler, küçük moleküllü bağlayıcıları tahmin etmek için evrime dayalı ligand bağlayıcı bilgileri kullanır [20] [21] ve hem küresel yapısal benzerliği hem de cep benzerliğini kullanabilir. [20] Küresel yapısal benzerlik temelli bir yaklaşım, PDB holo in şablon kütüphanesinde proteinlerle yapısal benzerlik bulmak için hem deneysel bir yapı hem de tahmin edilen bir protein modeli kullanır. Önemli yapısal benzerlik tespit edildikten sonra, seçilen holo PDB şablonlarından ekstrakte edilen ligandlara benzer küçük moleküller için taramaya 2D parmak izi tabanlı Tanimoto katsayı metriği uygulanır. [22] [23] Bu yöntemin tahminleri deneysel olarak değerlendirilmiştir ve aktif küçük moleküllerin tanımlanmasında iyi zenginleşme göstermektedir.
Yukarıda belirtilen yöntem, küresel yapısal benzerliğe bağlıdır ve ilgili proteinde belirli bir ligand bağlayıcı bölgeyi önceden seçemez . Ayrıca, yöntemler ligandlar için 2D benzerlik değerlendirmesine dayandığından, büyük ölçüde farklı olan ancak geometrik şekil benzerliği gösteren küçük moleküllerin stereokimyasal benzerliğini tanıyamazlar. Bu endişeleri gidermek için, holo protein şablonlarında spesifik bağlayıcı cepleri hedefleyebilen yeni bir cep merkezli yaklaşım olan PoLi geliştirildi ve deneysel olarak değerlendirildi.
BİLGİ İŞLEM ALTYAPISI
Birçok sanal tarama programının çalışması için bir ön koşul olan atomlar arasındaki ikili etkileşimlerin hesaplanması,hesaplama karmaşıklığı, burada N sistemdeki atom sayısıdır. Atom sayısına göre kuadratik ölçekleme nedeniyle, hesaplama altyapısı, ligand tabanlı bir yöntem için bir dizüstü bilgisayardan yapıya dayalı bir yöntem için bir ana bilgisayara kadar değişebilir.
LİGAND TABANLI
Ligand bazlı yöntemler tipik olarak tek bir yapı karşılaştırma işlemi için bir saniyenin bir kısmını gerektirir. Birkaç saat içinde büyük bir tarama yapmak için tek bir CPU yeterlidir. Bununla birlikte, büyük bir bileşik veri tabanının işlenmesini hızlandırmak için paralel olarak birkaç karşılaştırma yapılabilir.
YAPI TABANLI
Görevin boyutu, Sun Grid Engine veya Torque PBS gibi işi yürütmek için bir toplu kuyruk işlemcisi çalıştıran bir Linux sistemleri kümesi gibi paralel bir bilgi işlem altyapısı gerektirir .
Büyük bileşik kütüphanelerinden gelen girdileri işlemek için bir araç gereklidir. Bu, çeşitli hesaplama düğümlerine paralel olarak bileşikler ileterek, paralel küme tarafından sorgulanabilen bir bileşik veritabanı formu gerektirir. Ticari veritabanı motorları çok ağır olabilir ve Berkeley DB gibi yüksek hızlı bir indeksleme motoru daha iyi bir seçim olabilir. Ayrıca, iş başına bir karşılaştırma yapmak etkili olmayabilir, çünkü küme düğümlerinin hızlanma süresi yararlı iş miktarını kolayca geçebilir. Bu sorunu çözmek için, her küme işindeki bileşik gruplarını işlemek ve sonuçları bir tür günlük dosyasına toplamak gerekir. Günlük dosyalarını madencilik ve yüksek puanlama adayları çıkarmak için ikincil bir süreç daha sonra tüm deney çalıştırıldıktan sonra çalıştırılabilir.
DOĞRULUK
Sanal taramanın amacı, ilgili makromoleküler hedefe bağlanan yeni kimyasal yapı moleküllerini tanımlamaktır . Böylece, sanal bir ekranın başarısı, toplam isabet sayısından ziyade ilginç yeni iskeleler bulma olarak tanımlanır. Bu nedenle, sanal tarama doğruluğunun yorumları dikkatle ele alınmalıdır. İlginç iskele düşük isabet oranları zaten bilinen iskele yüksek isabet oranları üzerinde açıkça tercih edilir.
Literatürdeki sanal tarama çalışmalarının çoğu testi geriye dönüktür. Bu çalışmalarda, bir VS tekniğinin performansı, daha önce bilinen molekülleri, hedefin (aktif moleküller veya sadece aktifler) çok daha yüksek bir oranda varsayılan aktif olmayan madde içeren bir kitaplıktan yakınlığı ile alma kabiliyeti ile ölçülür. tuzağı. Buna karşılık, sanal tarama prospektif uygulamalarda ortaya çıkan hit deneysel teyit (örn maruz IC 50 ölçüm). Retrospektif kriterlerin prospektif performans için iyi bir belirleyici olmadığı ve sonuç olarak sadece prospektif çalışmaların, bir tekniğin belirli bir hedef için uygunluğunun kesin bir kanıtı olduğu konusunda fikir birliği vardır. [24] [25] [26][27] [28]
İLAÇ KEŞFİNE BAŞVURU
Sanal tarama, isabet moleküllerinin tıbbi kimya için bir başlangıç olarak tanımlanması söz konusu olduğunda çok yararlı bir uygulamadır. Sanal tarama yaklaşımı, tıbbi kimya endüstrisinde daha hayati ve önemli bir teknik olmaya başladığında, yaklaşım hızla artmıştır. [29]
LİGAND TABANLI YÖNTEMLER
Ligandların reseptöre nasıl bağlanacağını tahmin etmeye çalışan yapıyı bilmemekle birlikte. Farmakofor özellikleri ile her ligand tanımlanmış donör ve alıcıları özellikleri. Eşitlik özellikleri üst üste bindirilir, ancak olası tek bir doğru çözüm olmadığı göz önüne alındığında. [1]
FARMAKOFOR MODELLERİ
Bu teknik, referans bileşikleri, aynı tanımlayıcılar ve katsayı, fakat farklı aktif bileşikler kullanılarak arama sonuçlarının birleştirilmesinde kullanılır. Bu teknik faydalıdır, çünkü çeşitli aktifler söz konusu olduğunda en doğru performansın yanı sıra tek bir referans yapısı kullanmaktan daha verimlidir. [1]
Farmakofor, biyolojik tepkisini hızlandırmak için optimal bir molekülerüstü etkileşime veya biyolojik bir hedef yapı ile etkileşime sahip olmak için gerekli olan sterik ve elektronik özelliklerin bir grubudur. Bir aktifler kümesi olarak bir temsilci seçin, çoğu yöntem benzer bağlar arayacaktır. Birden fazla rijit molekülün olması tercih edilir ve ligandlar çeşitlendirilmelidir, başka bir deyişle bağlanma fazında meydana gelmeyen farklı özelliklere sahip olmalarını sağlar. [1]
YAPI
Bilinen aktif ve bilinen etkin olmayan bilgilere dayanarak bileşik bir tahmin modeli oluşturun. Küçük bir homojen veri kümesiyle sınırlı olan QSAR’ın (Nicel-Yapı Aktivite İlişkisi). Verilerin niteliksel olarak ele alındığı ve yapısal sınıflar ve birden fazla bağlayıcı mod ile kullanılabildiği SAR’lar (Yapı Aktivite İlişkisi). Modeller kurşun keşfi için bileşiklere öncelik verir. [1]
MAKİNE ÖĞRENİMİ
Makine Öğreniminin bu Sanal Tarama modeli için kullanılabilmesi için bilinen aktif ve bilinen aktif olmayan bileşiklere sahip bir eğitim seti olmalıdır. Daha sonra alt yapı analizi, özyinelemeli bölümleme, destek vektör makineleri, en yakın komşular ve sinir ağları aracılığıyla hesaplanan bir aktivite modeli de vardır. Son adım, bir bileşiğin aktif olma olasılığını bulmak ve daha sonra her bir bileşiği aktif olma olasılığına göre sıralamaktır. [1]
MAKİNE ÖĞRENİMİNDE ALTYAPI ANALİZİ
Büyük veri kümelerinde kullanılan ilk Makine Öğrenimi modeli, 1973’te oluşturulan Altyapı Analizi’dir. Her parça altyapısı, sürekli bir katkıyı belirli türde bir etkinlik haline getirir. [1] Altyapı, ilaç tasarımındaki yapıların analiz edilmesi söz konusu olduğunda büyük boyutsallığın zorluğunun üstesinden gelen bir yöntemdir. Çok seviyeli bir bina veya kuleye benzer yapılar için verimli bir altyapı analizi kullanılır. Geometri, başlangıçta ve doruğa doğru belirli bir yapı için sınır eklemlerini numaralandırmak için kullanılır. Özel statik yoğuşma ve sübstitüsyon rutinleri geliştirildiğinde, bu yöntemin önceki altyapı analiz modellerinden daha verimli olduğu kanıtlanmıştır. [30]
ÖZYİNELEMELİ BÖLÜMLEME
Yinelemeli bölümleme, nitel veriler kullanarak bir karar ağacı oluşturan yöntemdir. Kuralların, sınıfları düşük bir yanlış sınıflandırma hatasıyla parçalama şeklini anlamak ve hiçbir mantık bölünmesi bulunmayana kadar her adımı tekrarlamak Bununla birlikte, özyinelemeli bölümleme potansiyel olarak aynı oranda iyi modeller yaratabilme yeteneğine sahip olmayabilir. [1]
PROTEİN LİGAND KENETLENMESİ BİLİNEN YAPIYA DAYALI YÖNTEMLER
Ligand, bir öncelikli sıralama verirken tek bir ligandın en olası nedenini belirlemek için bir yerleştirme arama algoritması ve puanlama işlevi kullanarak bir protein içindeki aktif bir bölgeye bağlanabilir. [1] [31]
KAYNAKÇA
- Gillet V (2013). “Ligand Tabanlı ve Yapı Tabanlı Sanal Tarama” (PDF). Sheffield Üniversitesi.
- Rester U (Temmuz 2008). “Sanallıktan gerçeğe – Potansiyel müşteri keşfi ve olası müşteri optimizasyonu: tıbbi kimya perspektifi”. İlaç Keşfi ve Geliştirilmesinde Güncel Görüş . 11 (4): 559-68. PMID 18600572 .
- Rollinger JM, Stuppner H, Langer T (2008). Biyoaktif doğal ürünlerin keşfi için sanal tarama . İlaç Araştırmalarında İlerleme. Fortschritte der Arzneimittelforschung. Progres des Recherches Pharmaceuticaliques . İlaç Araştırmalarında İlerleme. 65 . sayfa 211, 213-49. doi : 10.1007 / 978-3-7643-8117-2_6 . ISBN 978-3-7643-8098-4. PMID 18084917 .
- Walters WP, Stahl MT, Murcko MA (1998). Msgstr “Sanal tarama – genel bakış”. Uyuşturucu Keşfi. Bugün . 3 (4): 160-178. doi : 10.1016 / S1359-6446 (97) 01163-X .
- Bohacek RS, McMartin C, Guida WC (1996). “Yapıya dayalı ilaç tasarımı sanatı ve uygulaması: moleküler modelleme perspektifi”. Med. Res. Rev . 16 (1): 3-50. doi : 10.1002 / (SICI) 1098-1128 (199601) 16: 1 <3 :: AID-MED1> 3.0.CO; 2-6 . PMID 8788213 .
- McGregor MJ, Luo Z, Jiang X (11 Haziran 2007). “Bölüm 3: İlaç keşfinde sanal tarama”. Huang Z’de (ed.). İlaç Keşfi Araştırması. Post-Genomik Dönemde Yeni Sınırlar . Wiley-VCH: Weinheim, Almanya. sayfa 63-88. ISBN 978-0-471-67200-5.
- McInnes C (Ekim 2007). “İlaç keşfinde sanal tarama stratejileri”. Kimyasal Biyolojide Güncel Görüş . 11 (5): 494-502. doi : 10.1016 / j.cbpa.2007.08.033 . PMID 17936059 .
- Sun H (2008). “Farmakofor tabanlı sanal tarama”. Güncel Tıbbi Kimya . 15 (10): 1018-24’te açıklanmaktadır. doi : 10.2174 / 092986708784049630 . PMID 18393859 .
KAYNAKÇA
- Willet P, Barnard JM, Downs GM (1998). “Kimyasal benzerlik araştırması”. Kimyasal Bilgi ve Bilgisayar Bilimleri Dergisi . 38 (6): 983-996. CiteSeerX 10.1.1.453.1788 . doi : 10.1021 / ci9800211 .
- Rush TS, Grant JA, Mosyak L, Nicholls A (Mart 2005). “Şekle dayalı 3-B iskele atlama yöntemi ve bakteriyel protein-protein etkileşimine uygulanması”. Tıbbi Kimya Dergisi . 48 (5): 1489-95. CiteSeerX 10.1.1.455.4728 . doi : 10.1021 / jm040163o . PMID 15743191.
- Ballester PJ, Westwood I, Laurieri N, Sim E, Richards WG (Şubat 2010). “Ultrafast Şekil Tanıma ile prospektif sanal tarama: yeni arilamin N-asetiltransferaz inhibitörlerinin tanımlanması” . Kraliyet Cemiyeti Dergisi, Arayüz . 7 (43): 335-42. doi : 10.1098 / rsif.2009.0170 . PMC 2842611 . PMID 19586957 .
- Kumar A, Zhang KY (2018). “Şekil Benzerlik Yöntemlerinin Geliştirilmesindeki Gelişmeler ve İlaç Keşfindeki Uygulamaları” . Kimyada Sınırlar . 6 : 315. Bibcode : 2018 FrCh …. 6..315K . doi : 10.3389 / fchem.2018.00315 . PMC 6068280 . PMID 30090808 .
- Li H, Leung KS, Wong MH, Ballester PJ (Temmuz 2016). “USR-VS: ultra hızlı şekil tanıma tekniklerini kullanarak büyük ölçekli ileriye dönük sanal tarama için bir web sunucusu” . Nükleik Asit Araştırmaları . 44 (W1): W436-41. doi : 10.1093 / nar / gkw320 . PMC 4987897 . PMID 27106057 .
- Sperandio O, Petitjean M, Tuffery P (Temmuz 2009). “wwLigCSRre: isabet tanımlama ve optimizasyon için 3D ligand tabanlı bir sunucu” . Nükleik Asit Araştırmaları . 37 (Web Sunucusu sorunu): W504–9. doi : 10.1093 / nar / gkp324 . PMC 2703967 . PMID 19429687 .
- Kroemer RT (Ağustos 2007). “Yapıya dayalı ilaç tasarımı: yerleştirme ve puanlama”. Güncel Protein ve Peptit Bilimi . 8 (4): 312-28. CiteSeerX 10.1.1.225.959 . doi : 10.2174 / 138920307781369382 . PMID 17696866 .
- Cavasotto CN, Orry AJ (2007). “Ligand yerleştirme ve ilaç keşfinde yapı tabanlı sanal tarama”. Tıbbi Kimyada Güncel Konular . 7 (10): 1006-14. doi : 10.2174 / 156802607780906753 . PMID 17508934 .
KAYNAKÇA
- Kooistra AJ, Vischer HF, McNaught-Flores D, Leurs R, de Esch IJ, de Graaf C (2016). “Kombine bir puanlama yöntemi kullanılarak GPCR ligandları için işleve özgü sanal tarama” . Bilimsel Raporlar . 6 : 28288. Bibcode : 2016NatSR … 628288K . doi : 10.1038 / srep28288 . PMC 4919634 . PMID 27339552 .
- Irwin JJ, Shoichet BK, Mysinger MM, Huang N, Colizzi F, Wassam P, Cao Y (Eylül 2009). “Otomatik yerleştirme ekranları: bir fizibilite çalışması” . Tıbbi Kimya Dergisi . 52 (18): 5712-20. doi : 10.1021 / jm9006966 . PMC 2745826 . PMID 19719084 .
- Li H, Leung KS, Ballester PJ, Wong MH (2014-01-24). “istar: büyük ölçekli protein-ligand yerleştirme için bir web platformu” . BİRİNCİ PLOS . 9 (1): e85678. Bibcode : 2014PLoSO … 985678L . doi : 10.1371 / günlük.pone.0085678 . PMC 3901662 . PMID 24475049 .
- Zhou H, Skolnick J (Ocak 2013). “FINDSITE (tarak): diş açma / yapıya dayalı, proteomik ölçekli sanal ligand tarama yaklaşımı”. Kimyasal Bilgi ve Modelleme Dergisi. 53(1): 230-40. doi:10.1021 / ci300510n. PMC 3557555 . PMID 23240691.
- Roy A, Skolnick J (Şubat 2015). “LIGSIFT: ligand yapısal hizalama ve sanal tarama için açık kaynaklı bir araçtır” . Biyoinformatik . 31 (4): 539-44. doi : 10.1093 / biyoinformatik / btu692 . PMC 4325547 . PMID 25336501 .
- Gaulton A, Bellis LJ, Bento AP, Chambers J, Davies M, Hersey A, Hafif Y, McGlinchey S, Michalovich D, Al-Lazikani B, Overington JP (Ocak 2012). “ChEMBL: ilaç keşfi için büyük ölçekli bir biyoaktivite veritabanı” . Nükleik Asit Araştırmaları . 40 (Veritabanı sorunu): D1100–7. doi : 10.1093 / nar / gkr777 . PMC 3245175 . PMID 21948594 .
- Wishart DS, Knox C, Guo AC, Shrivastava S, Hassanali M, Stothard P, Chang Z, Woolsey J (Ocak 2006). “DrugBank: silico ilaç keşfi ve keşif için kapsamlı bir kaynak” . Nükleik Asit Araştırmaları . 34 (Veritabanı sorunu): D668-72. doi : 10.1093 / nar / gkj067 . PMC 1347430 . PMID 16381955 .
- Wallach I, Heifets A (2018). “Ligand tabanlı sınıflandırma ölçütlerinin çoğu genelleme yerine ezberlemeyi ödüllendirir”. Kimyasal Bilgi ve Modelleme Dergisi . 58 (5): 916-932’de açıklanmaktadır. arXiv : 1706.06619 . doi : 10.1021 / acs.jcim.7b00403 . PMID 29698607 .
KAYNAKÇA
- Irwin JJ (2008). Msgstr “Sanal tarama için topluluk kriterleri” . Bilgisayar Destekli Moleküler Tasarım Dergisi . 22 (3-4): 193–9. Bibcode : 2008 JCAM.2.2..193I . doi : 10.1007 / s10822-008-9189-4 . PMID 18273555 .
- İyi AC, Oprea TI (2008). “CAMD tekniklerinin optimizasyonu 3. Sanal tarama zenginleştirme çalışmaları: takım seçiminde bir yardım mı yoksa engel mi?” . Bilgisayar Destekli Moleküler Tasarım Dergisi . 22 (3-4): 169-78. Önlük kodu : 2008JCAMD..22..169G . doi : 10.1007 / s10822-007-9167-2 . PMID 18188508 .
- Schneider G (Nisan 2010). “Sanal tarama: sonsuz bir merdiven?” . Doğa Yorumlar. İlaç Keşfi . 9 (4): 273-6. doi : 10.1038 / nrd3139 . PMID 20357802 .
- Ballester PJ (Ocak 2011). Msgstr “Ultrafast şekil tanıma: yöntem ve uygulamalar”. Gelecekteki Tıbbi Kimya . 3 (1): 65-78. doi : 10.4155 / fmc.10.280 . PMID 21428826 .
- Lavecchia A, Di Giovanni C (2013). “İlaç keşfinde sanal tarama stratejileri: eleştirel bir inceleme”. Güncel Tıbbi Kimya . 20 (23): 2839-60. doi : 10.2174 / 09298673113209990001 . PMID 23651302 .
- Gurujee CS, Deshpande VL (Şubat 1978). “Geliştirilmiş bir altyapı analizi yöntemi”. Bilgisayarlar ve Yapılar . 8 (1): 147-152. doi : 10.1016 / 0045-7949 (78) 90171-2 .
- Pradeepkiran, Jangampalli Adi; Reddy, P.Hemachandra (Mart 2019). “Alzheimer Hastalığında Fosforlu Tau İnhibitörleri için Yapı Tabanlı Tasarım ve Moleküler Yerleştirme Çalışmaları” . Hücreler . 8 (3): 260. doi : 10.3390 / hücreler8030260 . PMC 6468864. PMID 30893872 .